上一篇
f snp数据库
- 行业动态
- 2025-05-03
- 2869
F-SNP数据库是收录功能相关单核苷酸多态性的专业 数据库,旨在为基因功能研究、疾病关联分析等提供关键变异位点的
常见SNP数据库
以下是科研中常用的SNP数据库及其核心特点:
| 数据库名称 | 所属机构/项目 | 主要功能 |
|---|---|---|
| dbSNP | NCBI | 收录全球已知的人类及其他物种的SNP,提供基础变异信息(位置、等位基因)。 |
| gnomAD | Broad Institute & 国际合作团队 | 基于大规模人群测序数据,提供SNP的等位基因频率(AFR、AMR、EAS、EUR、SAS等)。 |
| ClinVar | NIH | 聚焦临床相关的SNP,记录变异与疾病的关联及临床意义解读。 |
| 1000 Genomes Project | 国际协作组 | 提供多人群基因组数据,包含SNP及其连锁信息,支持群体遗传学分析。 |
| Ensembl | EMBL-EBI | 整合基因注释与SNP功能预测(如编码区、调控区域),支持变异对基因功能的影响分析。 |
核心数据字段与功能
基础信息
- 染色体位置:chr:position(如chr7:140453136)
- 参考与替代等位基因:如rs12345678(A/G)
- 命名规则:以“rs”开头的唯一ID(如rs12345678)。
人群遗传学数据
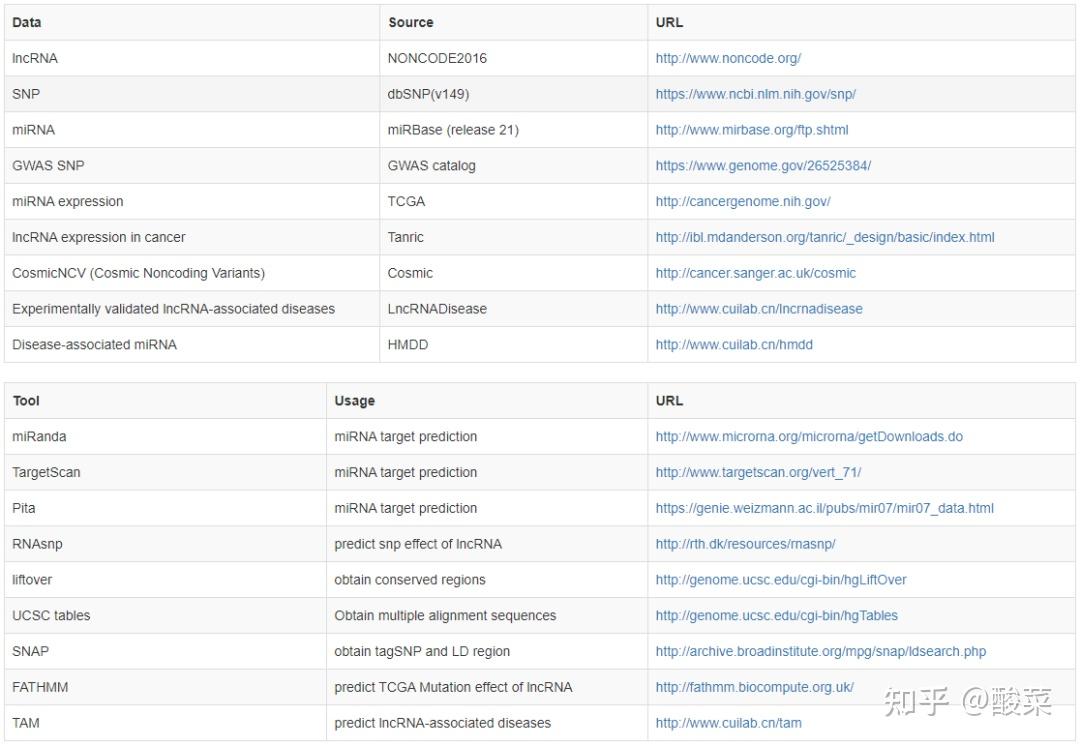
- 等位基因频率(AF):不同人群中的A/T/G/C比例。
- 连锁不平衡(LD):SNP间相关性,用于标签SNP选择。
功能注释
- 基因区域:编码区(Exonic)、剪接区(Splicing)、调控元件(如启动子)。
- 氨基酸改变:如p.Gly123Ser(蛋白质序列影响)。
- 致病性预测:SIFT、PolyPhen等工具评估功能危害。
疾病关联
- GWAS结果:SNP与复杂疾病的统计显著性(如P值、OR值)。
- 孟德尔病:HGMD数据库记录单基因病致病突变。
应用场景与工具推荐
| 研究需求 | 推荐数据库/工具 | 示例用途 |
|---|---|---|
| 查询SNP人群频率 | gnomAD、ALFA | 判断罕见变异(MAF<0.01%)或常见变异。 |
| 分析功能影响 | Ensembl Variant Effect Predictor | 预测SNP是否导致蛋白失活或剪接异常。 |
| 疾病关联研究 | ClinVar、GWAS Catalog | 查找已报道的致病突变或疾病风险位点。 |
| 进化保守性分析 | PhastCons(UCSC Genome Browser) | 评估SNP所在区域的进化保守性,推测功能重要性。 |
访问方式与数据下载
- 网页检索:通过关键词(如基因名、rsID)、染色体位置筛选。
- API接口:程序化批量获取数据(如Ensembl BioMart、dbSNP FTP)。
- 格式:支持VCF、TSV、JSON等格式,便于生物信息学工具分析。
相关问题与解答
问题1:如何判断一个SNP是否具有潜在致病性?
解答:
- 人群频率:若SNP在对照人群中频率极低(如gnomAD MAF<0.001%),可能为罕见致病变异。
- 功能预测:使用SIFT/PolyPhen评估是否影响蛋白功能,或通过Ensembl查看是否位于关键调控区域。
- 疾病关联:在ClinVar中检索该SNP是否被标注为致病/良性,或在GWAS Catalog中查找关联研究。
问题2:dbSNP与gnomAD的数据有什么区别?
解答:
| 维度 | dbSNP | gnomAD |
|————————|——————————–|————————————-|
| 数据来源 | 文献提交、测序项目汇总 | 大规模人群测序(141,456个样本) |
| | SNP基础信息(位置、等位基因) | 人群等位基因频率、功能注释、LD数据 |
| 适用场景 | 变异初步鉴定 | 人群遗传背景分析、罕见病研究 |
如需进一步分析特定SNP,可结合多个数据库交叉验证功能与疾病