上一篇
如何利用GATK优化测序数据分析结果并提升准确率?
- 行业动态
- 2025-04-26
- 3634
GATK(Genome Analysis Toolkit)是一套广泛使用的基因组数据分析工具,专注于变异检测(如SNP、Indel),其流程涵盖数据预处理、变异识别及结果优化,支持全基因组和外显子组测序,遵循最佳实践确保准确性,广泛应用于科研与临床研究中高通量测序数据的变异筛选。
基因组测序数据的分析是精准医学和生物研究的核心环节之一,而GATK(Genome Analysis Toolkit)作为行业标杆工具,被广泛应用于从原始测序数据到变异位点发现的完整流程,以下内容将从原理、流程、应用场景及注意事项展开,为访客提供全面且实用的技术解析。
GATK的核心功能与原理
GATK由Broad Institute开发,专为高通量测序数据分析设计,尤其擅长处理人类全基因组和外显子组数据,其核心算法基于贝叶斯统计模型,通过比对测序数据的覆盖深度、碱基质量值等信息,精准识别单核苷酸多态性(SNP)、插入缺失(Indel)等变异。
- 关键模块:
- 数据预处理:去除低质量序列(Base Quality Score Recalibration, BQSR)和重复序列(MarkDuplicates)。
- 变异检测:HaplotypeCaller算法通过局部重组比对(Local Assembly)提高复杂区域检测准确率。
- 变异过滤:使用VQSR(Variant Quality Score Recalibration)或硬过滤(Hard Filtering)剔除假阳性结果。
- 功能注释:结合ClinVar、dbSNP等数据库,预测变异致病性。
标准分析流程(以WGS为例)
原始数据质控
使用FastQC评估原始测序文件(FASTQ)的质量,通过Trimmomatic或Cutadapt去除接头和低质量碱基。比对与排序
利用BWA-MEM将reads比对到参考基因组(如GRCh38),输出BAM文件;通过Picard或samtools排序和标记重复序列。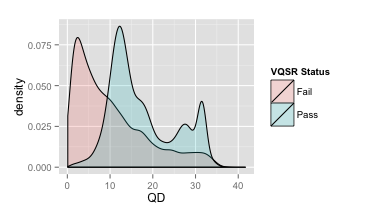
变异检测
- 单样本分析:
HaplotypeCaller生成GVCF文件。 - 多样本联合分析:
GenotypeGVCFs整合样本数据,生成VCF格式的变异集合。
- 单样本分析:
变异过滤与注释
- 应用VQSR模型(需已知变异数据库如HapMap和1000 Genomes)进行机器学习筛选。
- 使用SnpEff或ANNOVAR注释变异的基因功能及临床意义。
适用场景与注意事项
典型应用
- 临床诊断:检测癌症体细胞突变或遗传病致病位点。
- 群体遗传学:研究人群结构、自然选择信号。
- 农业基因组学:筛选作物/家畜的优良性状关联变异。
关键问题
数据质量要求
- 建议全基因组测序深度≥30×,外显子组≥100×。
- 若覆盖深度不足,可能遗漏低频变异。
计算资源优化
- GATK 4.x支持Spark分布式计算,可缩短大规模数据分析时间。
- 推荐使用高性能计算集群(至少64GB内存,16核CPU)。
版本与参数选择
不同版本(如GATK 3.8 vs. 4.3)的算法差异可能影响结果,建议参考官方Best Practices文档调整参数。
常见问题解答(FAQ)
Q1:GATK能否用于非人类物种?
是的,但需提供对应物种的参考基因组和已知变异数据库(若无,需关闭VQSR,改用硬过滤)。
Q2:如何验证分析结果的可靠性?
- 使用标准品系(如NA12878)验证检测灵敏度与特异性。
- 交叉比对多个变异检测工具(如GATK vs. Strelka)。
Q3:GATK流程的耗时与成本?
全基因组分析约需48-72小时(取决于样本量和硬件配置),建议云服务器按需付费模式降低成本。
权威参考与工具
- GATK官方文档:
https://gatk.broadinstitute.org - 千人基因组计划数据库:
https://www.internationalgenome.org - 文献支持:
DePristo, M. A. et al. (2011). A framework for variation discovery and genotyping using next-generation DNA sequencing data. Nature Genetics, 43(5), 491-498.