上一篇
silva数据库怎么比对
- 数据库
- 2025-07-23
- 4
SILVA数据库比对需先下载适配版本(如123版),通过ARB或mothur软件匹配rRNA序列至对应亚基(如16S/23S)进行分类鉴定
Silva数据库(全称SILVA ribosomal RNA database)是一个综合性的核糖体RNA(rRNA)基因序列数据库,专注于提供高质量、经过人工校验的SSU(小亚基,如16S/18S rRNA)和LSU(大亚基,如23S/28S rRNA)序列数据,其核心功能是为微生物生态学、系统发育分析等领域提供序列比对参考,帮助研究者准确分类和注释rRNA序列,以下是Silva数据库比对的详细流程和关键要点:
比对前的准备
-
数据库选择与下载
- 访问官网:Silva数据库官网(https://www.arb-silva.de/)提供多种数据下载选项,包括按版本号、序列类型(SSU/LSU)、区域(如细菌、古菌、真核生物)分类的数据集。
- 推荐下载内容:
- SSU rRNA序列(如
SILVA_138.1_SSU.fasta),适用于16S/18S比对; - LSU rRNA序列(如
SILVA_138.1_LSU.fasta),适用于23S/28S比对。
- SSU rRNA序列(如
- 版本选择:优先使用最新版本(如Silva v138.1),以确保序列数据的完整性和准确性。
-
序列格式处理
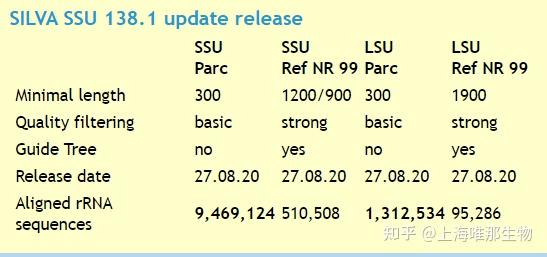
- U与T的转换:真核生物18S/28S rRNA序列中可能包含尿嘧啶(U),需统一转换为胸腺嘧啶(T)以匹配数据库格式。
- 合并参考序列:若需同时比对SSU和LSU,需将两者的序列文件合并为一个参考库,并删除重复序列。
比对工具与参数设置
Silva数据库支持多种比对工具,需根据研究需求选择:
| 工具 | 适用场景 | 关键参数 |
|---|---|---|
| BLAST(如blastn) | 快速筛选高相似性序列 | -evalue 0.001(控制误报率)、-max_target_seqs 5(限制输出结果数量)、-dust no(禁用复杂滤波) |
| Infernal(CMpress) | 精准识别保守的rRNA结构域 | 依赖Covariance Model(CM)模型,需配合Silva提供的SILVA_138.1_cm.tar.gz文件 |
| DRAM-GOB(SSU-specific) | 超高速16S/18S比对 | 直接使用默认参数,支持批量处理 |
比对流程示例(以BLAST为例)
-
建立本地数据库
makeblastdb -in SILVA_138.1_SSU.fasta -title Silva_SSU -dbtype nucl
-
运行比对
blastn -query input_sequences.fasta -db Silva_SSU -outfmt 6 -evalue 0.001 > results.txt
-
结果解析
- 高分位匹配:关注
bitscore > 200且e-value < 0.001的结果,通常对应物种水平的鉴定。 - 低分位匹配:若结果中出现
e-value偏高或相似度低于90%,可能是非目标rRNA或被墙序列,需结合其他数据库(如RDP)交叉验证。
- 高分位匹配:关注
常见问题与解决方案
FAQs
-
问题1:如何选择合适的Silva数据库版本?
- 解答:优先使用最新版本(如v138.1),因其包含更全面的序列和修正错误,若需复现历史研究,需下载对应版本的数据库(官网提供存档版本)。
-
问题2:比对结果中出现大量低相似度匹配,如何处理?
- 解答:
- 过滤
e-value > 0.001或similarity < 80%的结果; - 检查输入序列是否含非rRNA片段(如基因组DNA被墙),可先用
SortMeRNA等工具预过滤; - 尝试使用更严格的比对工具(如Infernal)或启用Silva的“质量过滤”功能。
- 过滤
- 解答:
通过以上流程,可高效利用Silva数据库进行rRNA序列比对,为下游的微生物分类、系统发育分析提供可靠